張弓研究組在《PLOS Genetics》發表翻譯組學突破性文章
2016-06
文章來自: 閱讀次數:497
2016年2月29日,國際著名學術雜志《PLOS genetics》上在線發表了太阳集团app首页生命與健康工程研究院張弓、王通、何慶瑜教授課題組一篇最新翻譯組學突破性研究成果,研究首次測定生理條件下所有基因的翻譯延伸速率,實現了多種技術創新,終結了多項學界長期争論的問題。
研究首次實現了生理條件下人細胞内所有基因的翻譯延伸速率測定,發現tRNA豐度是決定翻譯速率的幾乎唯一的因素,而傳統上用于預測翻譯速率的各項密碼子偏好性指數與實測的翻譯速率毫無關系。尤其是,該研究首次以實驗證據表明,癌細胞可減慢促癌基因的翻譯速率,進而保證這些基因産物的正确折疊,從而增強癌症表型;與之相反,癌細胞加速抑癌基因的翻譯速率使其難以正确折疊而削弱其功能,從而維持癌細胞的惡性表型。
翻譯調控是近年來被認為在生物信息流中具中樞地位。早在2001年,Pradet-Balade在綜述中就指出,對翻譯中mRNA的測定非常重要。2011年,Schwanh甀猀猀攀爀等人在Nature上發表論文,用數學模型證實翻譯調控占從基因組到蛋白質組所有調控的一半以上,超過其他所有調控方式(包括轉錄調控、mRNA降解調控、蛋白質降解調控)的總和,是當之無愧的調控之王。因此,對翻譯的全局研究顯得尤為重要。但多年來,由于翻譯組研究的技術困難,使得該領域的研究一直進展非常緩慢。2013年,太阳集团app首页張弓、王通、何慶瑜課題組首次成功實現翻譯組測序,對人細胞内翻譯中的mRNA進行了全長定量測序,并發現翻譯起始效率高的基因直接調控細胞特異性表型。
作為翻譯調控的另一方面,翻譯延伸速率的全局測定則更加困難。2011年,Jonathan Weissman等人使用ribosome profiling技術對小鼠胚胎幹細胞進行了分析,他們使用一種藥物阻止翻譯起始,然後在不同時間點進行ribosome profiling,分析核糖體在mRNA上移動留下的空白區域的延長,測定出所有基因平均的翻譯延伸速率約為5.6 codons/s。但該研究隻能測定大量基因的平均翻譯延伸速率,而無法實現單基因水平測定,而他們的結果顯示不同類型基因的平均延伸速率并沒有顯著差異。此外,他們使用了藥物阻止翻譯起始,這是對細胞的嚴重幹擾,會造成約60秒的延遲,因而觀察到的并非生理狀況下的翻譯延伸。由于這些問題,他們的這種方法實際意義并不大,此後也未有人利用這種方法對翻譯延伸進行系統研究。
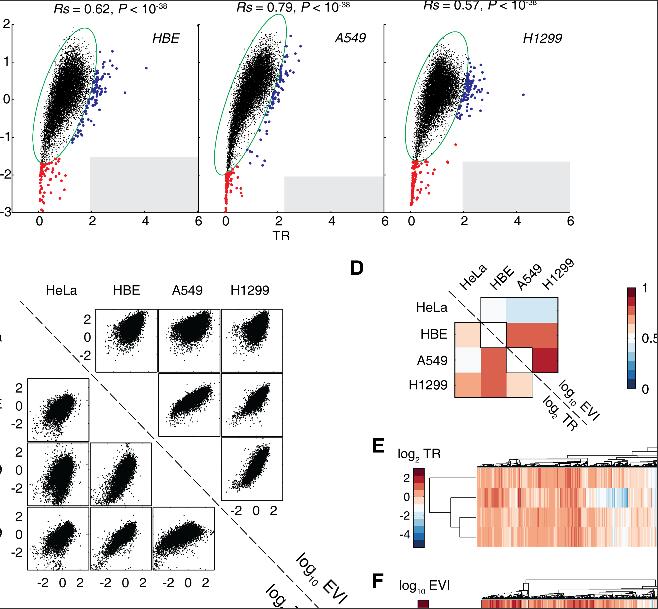
太阳集团app首页團隊則另辟蹊徑解決了這一問題。藉由其在全長翻譯組測序方面的技術優勢,他們對同一細胞系同時進行了mRNA測序(mrna-seq)、全長翻譯中mRNA測序(RNC-seq),以及核糖體足迹測序(ribosome profiling, Ribo-seq)。無需對細胞的正常生理狀态作任何人工幹預,這三大測序的數據可以直接計算出生理條件下每個基因的翻譯起始效率(以翻譯比例TR表征)和翻譯延伸速率(以延伸速率指數EVI表征)。使用課題組自行研發的超高精度序列比對算法FANSe2對測序數據進行處理,他們在4株細胞(HeLa, HBE, A549, H1299)中測定了8000-10000個基因的翻譯起始效率和EVI。
他們的研究結果表明,翻譯起始效率和翻譯延伸速率有一定的相關性,不存在翻譯起始效率很高但翻譯延伸很慢的基因,否則将會造成嚴重的核糖體“堵車”現象(ribosome jamming)而造成翻譯資源的無端浪費。
他們發現了另一有趣現象,即不同的細胞内,翻譯延伸減速的基因各不相同。這是什麼造成的?自上世紀80年代以來,mRNA的二級結構是否對翻譯延伸造成顯著影響一直是一個廣受争議的話題,正反雙方的論文都不少,然而基本都是單個基因的分析,并未有一個全局性的結論。暨大此項全基因組級别的研究可能為這場大辯論畫上一個句号:mRNA二級結構的穩定性對翻譯延伸速率在全局上沒有任何顯著影響。此前還有一種觀點認為,帶正電荷的氨基酸會減慢翻譯延伸速率,但此項研究證明,在多數細胞内,翻譯十分緩慢的基因所帶的正電荷氨基酸比例甚至要比全基因組的背景值還低,這推翻了此前的結論。在排除了衆多因素之後,研究者發現tRNA濃度介導的密碼子選擇是幾乎唯一的調控翻譯延伸速率的因素,并鑒定出了翻譯延伸緩慢基因所喜用的密碼子,它們也呈現出很強的細胞特異性。而幾十年來人們一直用來預測翻譯延伸速率的各種密碼子偏好指數,如codon adaptation index, codon bias index, tRNA adaptation index等與實測的翻譯延伸速率毫無關系,不應再使用。
同一組織來源的不同細胞系之間,翻譯延伸速率的調控是否有着生物學的意義?研究者對同樣來自于肺的正常細胞和癌細胞進行了分析,發現癌細胞降低了一些基因的翻譯速率,而這些基因的功能高度富集于癌症相關通路中。衆所周知,癌細胞中tRNA豐度較高,蛋白質折疊效率普遍較差,未折疊蛋白質顯著上升,然而這些翻譯延伸速率相對下調的基因的蛋白質産量和未正确折疊的蛋白質都沒有增加,證明下調翻譯速率能有效保證這些癌基因蛋白的折疊,從而使這些癌基因正常行使其功能。與之相反,已知的抑癌基因在癌細胞中的EVI被上調,加快翻譯延伸的結果是使抑癌基因難以正确折疊,從而難以發揮抑癌作用。這一正一反的翻譯延伸調控,使得癌細胞巧妙地選擇性地維持其惡性表型。
這項研究突破了翻譯組學的一大技術瓶頸,使得人們從此可以精确對翻譯延伸速率進行全局研究,并為翻譯延伸調控的生物學意義提供了全新的視角。
Genome-Wide and Experimental Resolution of Relative Translation Elongation Speed at Individual Gene Level in Human Cells
原文鍊接:DOI: 10.1371/journal.pgen.1005901
原文摘要:In the process of translation, ribosomes first assemble on mRNAs (translation initiation) and then translate along the mRNA (elongation) to synthesize proteins. Elongation pausing is deemed highly relevant to co-translational folding of nascent peptides and the functionality of protein products, which positioned the evaluation of elongation speed as one of the central questions in the field of translational control. By integrating three types of RNA-seq methods, we experimentally and computationally resolved elongation speed, with our proposed elongation velocity index (EVI), a relative measure at individual gene level and under physiological condition in human cells. We successfully distinguished slow-translating genes from the background translatome. We demonstrated that low-EVI genes encoded more stable proteins. We further identified cell-specific slow-translating codons, which might serve as a causal factor of elongation deceleration. As an example for the biological relevance, we showed that the relatively slow-translating genes tended to be associated with the maintenance of malignant phenotypes per pathway analyses. In conclusion, EVI opens a new view to understand why human cells tend to avoid simultaneously speeding up translation initiation and decelerating elongation, and the possible cancer relevance of translating low-EVI genes to gain better protein quality.